【保存版】今さら聞けない遺伝子発現制御ツールの違いと選択チャート

生命科学の実験では、遺伝子機能と表現型の関連を解析するための遺伝子発現制御が欠かせません。
それぞれのラボでさまざまな遺伝子発現制御ツールを使っているかと思いますが、既存のツールに使い慣れているからこそ、他のツールの最新情報を追いかけられていない、既存のツールで実験がうまくいかなかったときの他の選択肢がわからない、ということはないでしょうか。
本記事では、遺伝子発現制御ツールのうち代表的なRNA干渉(RNA interference, RNAi)とCRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)について解説します。
特にCRISPRはノックアウトだけでなく、ノックダウンと発現亢進にも応用でき、ライブラリーとして販売されているものもあります。遺伝子発現制御ツールの最新情報をキャッチして、みなさんの実験に役立ててください。
遺伝子発現制御の3パターン
遺伝子発現制御には、大きく分けるとノックダウンとノックアウト、そして発現亢進の3つがあります。
ノックダウンは、RNAiなどの手法を用いてターゲット遺伝子の発現量を減少させる手法です。次の解説するノックアウトでは致死となる遺伝子の機能解析に有効な場合があります。最近ではRNAiなどの手法以外にも、CRISPR技術を応用した方法も登場しています。
ノックアウトは、ターゲット遺伝子の発現を完全に消失させ、遺伝子機能を欠損させることです。以前は遺伝子組換え技術が必要で時間やコストの負担が大きかったのですが、CRISPR技術の登場により短時間で簡便にノックアウトできるようになっています。
発現亢進は、ターゲット遺伝子の発現量を増加させる手法です。過剰発現の方法もありますが、最近ではCRISPR技術を応用して転写レベルで発現亢進させる方法も登場しています。
主な遺伝子発現制御ツールのご紹介
RNAi(siRNA, esiRNA, shRNA)技術の基礎とその用途
RNAiは、転写後の遺伝子発現を制御する現象です。短い二本鎖RNAを用いてターゲットmRNAからタンパク質への翻訳を抑制します。完全に翻訳を阻害するわけではないので、RNAiは遺伝子発現を部分的に抑制するノックダウンに分類されます。主なRNAiには、siRNA, esiRNA, shRNAがあります(図1)。
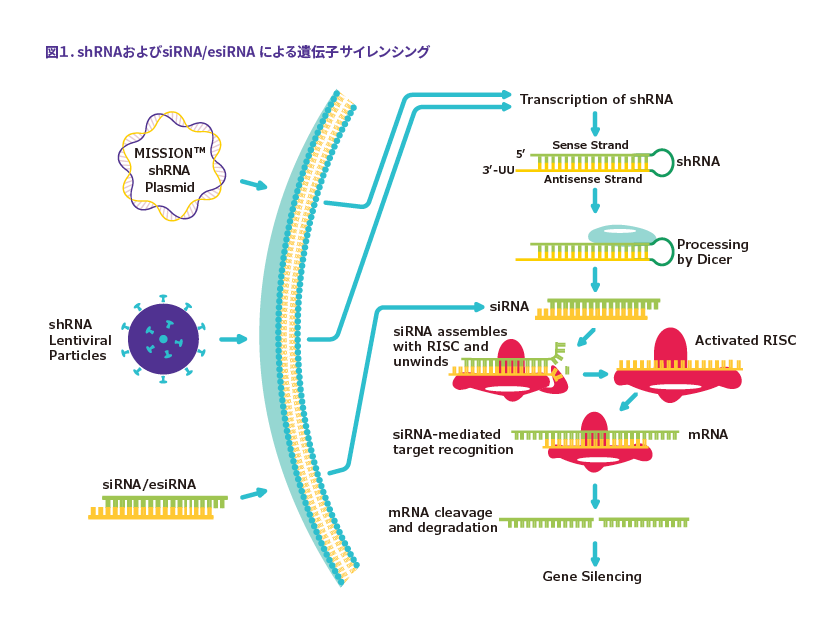
small interfering RNA(siRNA)は、人工的に合成した二本鎖RNAを細胞に導入します。細胞に導入されたsiRNAは、RNA-induced silencing complex(RISC)という複合体に取り込まれ、センス鎖のみが放出されます。RISCに取り込まれたままのアンチセンス鎖と相補的な配列をもつターゲットmRNAがRISCに取り込まれ、切断、分解されます。こうして翻訳を阻害します。
MISSION™ siRNAを使用したRNAi実験のポイント RNAi実験の基礎 siRNAのトランスフェクションプロトコール
siRNAは約20塩基対という短さゆえ、ターゲットmRNA以外にも作用してしまうオフターゲット効果(オフターゲット作用)のリスクがあります。オフターゲット効果のリスクを低減するために開発されたのがEndoribonuclease-prepared siRNA(esiRNA)です。
esiRNAは、300〜600塩基対をターゲット領域として、その二本鎖をエンドリボヌクレアーゼで処理して数百種類のsiRNAのプールとして構成したものです。個々のsiRNAは低濃度なのでオフターゲット効果を抑えつつ、効果的なノックダウンが期待できます。
small hairpin RNA(shRNA)は、図1に示すように細胞にプラスミドを導入またはレンチウイルスを感染させ、細胞内でshRNAを産生させます。shRNAはDicerというエンドヌクレアーゼによってヘアピン部分が切断され、siRNAが生成されます。ゲノムDNAに組み込まれれば、恒久的にノックダウンできます。
RNAi実験の基礎 shRNA レンチウイルスによるノックダウン RNAi実験の基礎 shRNA導入細胞を選択する抗生物質濃度の最適化
siRNA, esiRNA, shRNAの比較
siRNA、esiRNA、shRNAの比較を表1に示します。
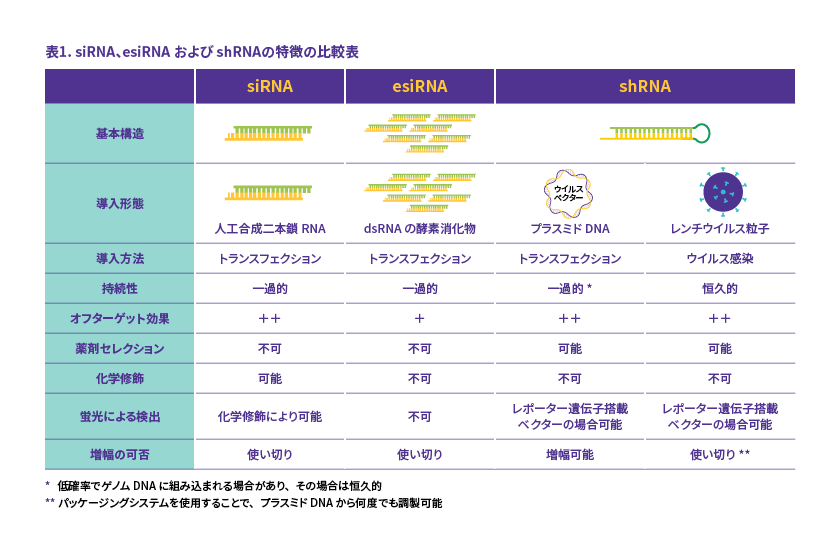
恒久的なノックダウンが目的の場合にはレンチウイルスによるshRNA、オフターゲット効果を抑制したいときはesiRNAがおすすめです。siRNAは人工的に合成する際に化学修飾を付加することで、siRNAの安定性を高めることや蛍光修飾で可視化することも可能です。
大量に実験を予定している場合には、ラボで増幅できるプラスミド形態にするとコストを抑えられるかもしれません。
実験の目的に応じて適切なツールを選択してください。
CRISPR(CRISPR-Cas9, CRISPRi, CRISPRa)技術の基礎とその用途
DNA配列を改変して遺伝子機能を欠損させるノックアウトを行うのがゲノム編集です。ゲノム編集の中でも簡便で安価なCRISPR-Cas9(クリスパー・キャスナイン)が現在のノックアウトツールの主流です。
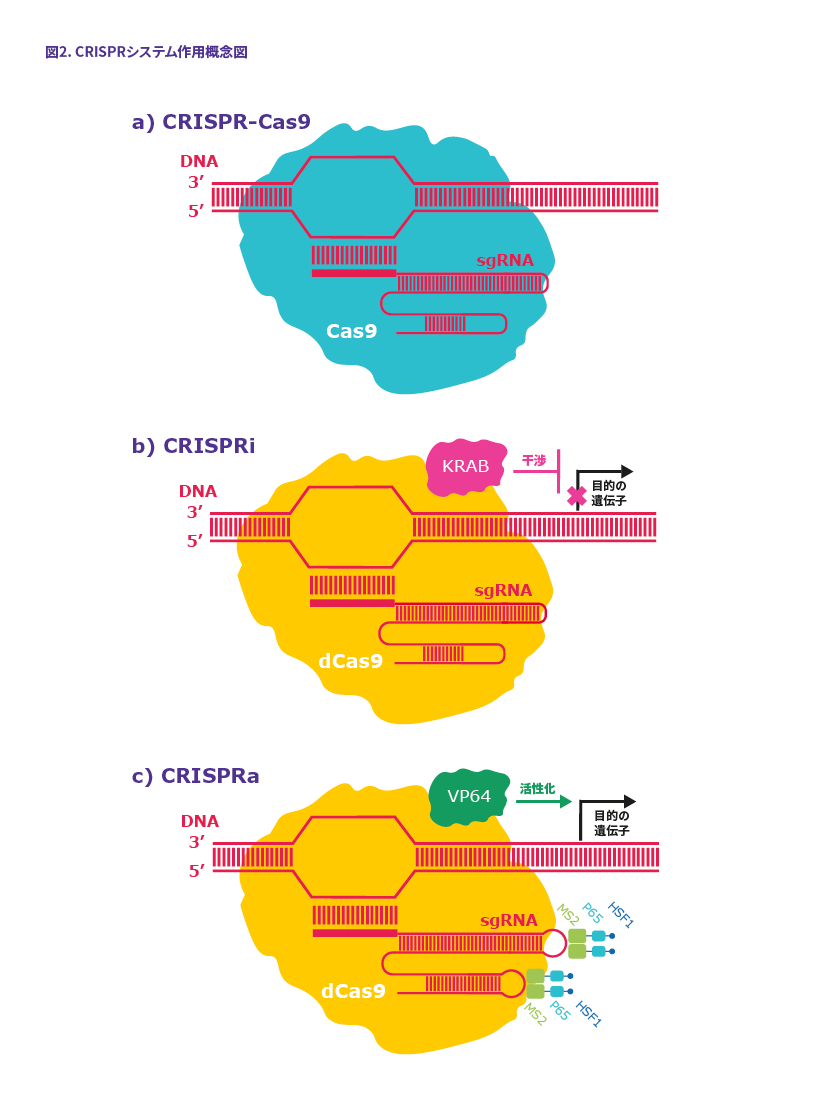
CRISPR-Cas9は、一本鎖のシングルガイドRNA(sgRNA)と、Cas9というDNAヌクレアーゼから構成されます。sgRNAがターゲットDNA配列に結合するとCas9を引き寄せ、Cas9がDNAの二本鎖を切断します。切断されたDNAを修復する際、エラーが発生してもとの配列とは異なる場合があります。この修復エラーを利用して遺伝子機能をノックアウトします(図2、a)。
CRISPR-Cas9といえばノックアウトという印象が強いかもしれませんが、DNAテンプレートを導入すればノックインも可能です。また、現在では、DNA切断活性を欠失させた変異型Cas9(dCas9)を用いてノックダウンまたは発現亢進も可能になったツールがあります。
ノックダウンでは、dCas9に、転写抑制因子であるKRABドメインを融合させたものを使用します。sgRNAがターゲットDNA配列に結合するとdCas9との複合体を形成し、KRABドメインによってターゲット遺伝子の転写が抑制されます。これをCRISPR interference(CRISPRi)とよびます(図2、b)(1)。
一方、発現亢進させるものがCRISPR activation (CRISPRa)です。CRISPRaでは、ウイルス由来の転写活性化因子であるVP64をdCas9に融合させたものを使用します。sgRNAにはMS2 RNAアプタマーが結合しており、転写因子により、遺伝子発現を亢進させる工夫も施されています(図2、c)(1, 2)。
シグマ アルドリッチのCRISPRiとCRISRPaの概要について
CRISPR-Cas9, CRISPRi, CRISPRaの比較
CRISPR-Cas9、 CRISPRi、CRISPRa、さらにRNAiと比較したものが表2です。
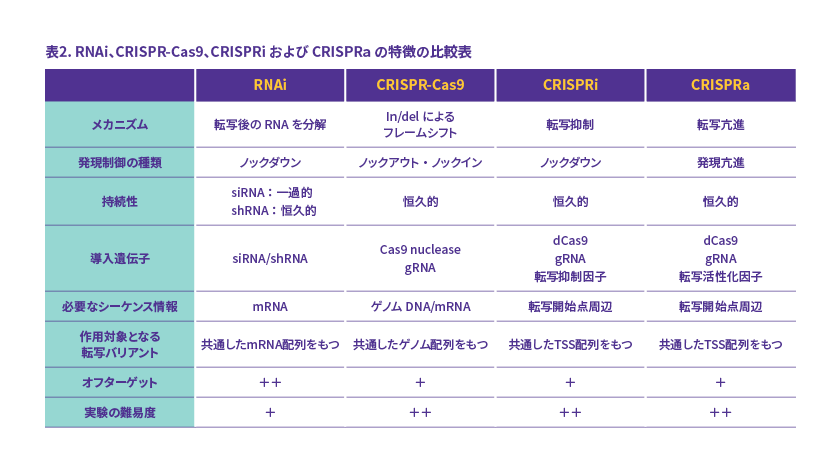
CRISPR-Cas9などはオフターゲット効果が比較的低いとされていますが、ゼロではありません。オフターゲット効果をさらに抑えたいときや多数のsgRNAを使用する場合には、sgRNAとCas9タンパク質の複合体を試験管内で調製してから細胞に導入するRNP法という方法もあります。
CRISPR-Cas9の導入方法(プロトコール)と応用例とは?
遺伝子発現制御ツールの選択チャート
今回紹介した遺伝子発現制御ツールの選択チャートを図3に示します。
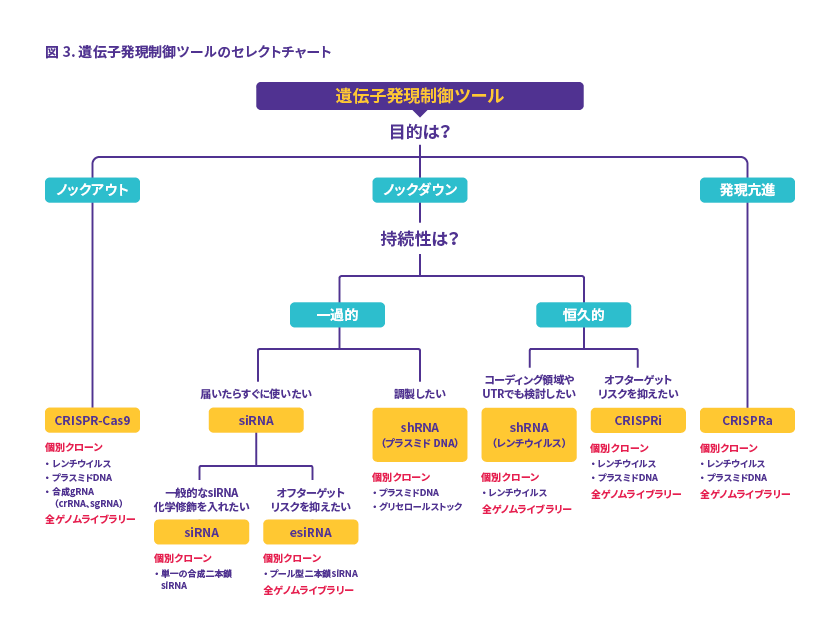
まず、実験目的がノックアウトか、ノックダウンか、発現亢進かで大きく分類します。目的がノックアウトならCRISPR-Cas9、発現亢進ならCRISPRaとなります。
ノックダウンが目的の場合、さらに持続性で分類します。恒久的にノックダウンしたい、かつオフターゲット効果を抑えたいのであればCRISPRiがおすすめです。ただし、CRISPRiはメカニズムの関係上、ターゲットは転写開始点付近のDNA配列に限定されます。コーディング領域やUTRをターゲットにする場合には、レンチウイルスを使用するshRNAもご検討ください。
一過的なノックダウンが目的ですぐに使用したい場合にはsiRNAが最適です。オフターゲット効果を抑えたいときにはesiRNAを選択します。
MISSION™ siRNAを使用したRNAi実験のポイント - M-hub(エムハブ)
新たな遺伝子発現制御ツールで実験の可能性を広げよう
この記事では、ノックダウンやノックアウト、発現亢進に適した遺伝子発現制御ツールを紹介しました。多くの製品で、個別クローンまたは全ゲノムライブラリーを用意しております。
shRNA libraryを使用したお客様の事例 CRISPR library —知っておきたい製品選定のコツ— CRISPR screeningとは?基礎やワークフロー、ライブラリー選択方法を解説
新たな情報を集めることで実験のアイデアが広がり、オフターゲット効果のようなトラブル解決にもつながるかもしれません。
メルクでは、RNAiやCRISPR関連の製品を幅広く用意しております。ご注文だけでなく、気軽なご相談にも対応します。
製品リストやご注文、お問い合わせ先などは以下のページに掲載しております。
・MISSION™ siRNA (対象製品一覧内RNAタブ参照) ・MISSION™ shRNA, MISSION™ CRISPR ・MISSION™ esiRNA
尚、遺伝子発現制御関連製品について、テクニカルスタッフによるご相談も承っています。
ぜひご活用ください。
<References>
1. Kendall R Sanson et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature communications, 9(1), 5416-5416 (2018-12-24)
2. Joung, J. et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nature Protocols. 12.4 (2017): 828-863.

下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須