CRISPR screening(CRISPRスクリーニング)とは?基礎やワークフロー、ライブラリー選択方法を解説

※2022年9月27日の記事を加筆・編集しました。
CRISPR-Cas9(クリスパー・キャスナイン)はゲノム編集ツールの中でも簡便で安価という特長があり、全ゲノム遺伝子ライブラリーによるスクリーニング(CRISPR screening)にも利用されています。
当記事では、CRISPR screeningの原理やワークフロー、ライブラリー選択方法などを解説します。特に培養細胞を使ったCRISPR screeningを検討する際にご参考にしてください。
CRISPR screeningとは?
CRISPR screeningの基本コンセプトは、CRISPR-Cas9による遺伝子ノックアウトをゲノムスケールで行うことで、特定の表現型に関連する遺伝子を包括的に検出することです。
CRISPR-Cas9とはゲノム編集技術の一種で、現在最も多く使われている手法です。CRISPR-Cas9は、標的とするDNA配列を特異的に認識して結合するガイドRNA(gRNA)と、ヌクレアーゼであるCas9から構成されます。詳しい原理や性質については、以下の記事をご覧ください。
CRISPR screeningでは、CRISPRライブラリーと呼ばれる多数の遺伝子をターゲットにしたgRNAを用意し、CRISPRライブラリーおよびCas9タンパク質を培養細胞に導入した後、薬剤耐性など目的に応じた化合物処理によるスクリーニングを行い、ノックアウトされた遺伝子を特定します。
CRISPR 登場以前から、ゲノムワイドな遺伝子スクリーニングにはshRNAライブラリーが利用されてきました。CRISPRスクリーニングでは、遺伝子を抑制ではなく完全に機能を欠失(ノックアウト)できる点が大きな特長です。また、遺伝子ノックアウトだけでなく、ウイルス由来の転写活性化因子VP64を利用した発現亢進(CRISPRa)、転写抑制因子であるKRABドメインを利用した発現抑制(CRISPRi)も可能です。
また、ゲノムスケールではなく、特定の酵素群*1や膜タンパク質*2などに限定したカスタムライブラリーを作成することで、費用を抑えつつサブゲノムスケールでCRISPR screeningを行う方法もあります。
弊社では様々なCRISPRライブラリーを取り揃えています。CRISPR screeningの製品ラインナップはこちらから、カスタマイズなど、詳細についてはご相談フォームからお問い合わせください。オンライン面談でもご説明可能です。
テクニカルスタッフに相談する
※オンライン技術サポートも実施中
CRISPR screeningの原理
CRISPR screeningに用いられるライブラリーは「プール型」と「アレイ型」の2種類があります*3。必要な設備やコスト、スクリーニング方法やリードアウトのオプションなどに大きな違いがあるので、以下にそれぞれの原理や利点、注意点を述べます。
プール型
1本または複数のチューブに多数のgRNAが含まれているタイプです。レンチウイルス粒子を作成し、細胞集団に対してgRNAを導入します(図1)。必要に応じて、gRNA/Cas9導入細胞を薬剤や蛍光マーカーなどでセレクションします。薬剤耐性などの目的の表現型スクリーニング後、次世代シーケンサー(NGS)を用いてゲノムに組み込まれたgRNA配列と表現型の関係を明らかにします。
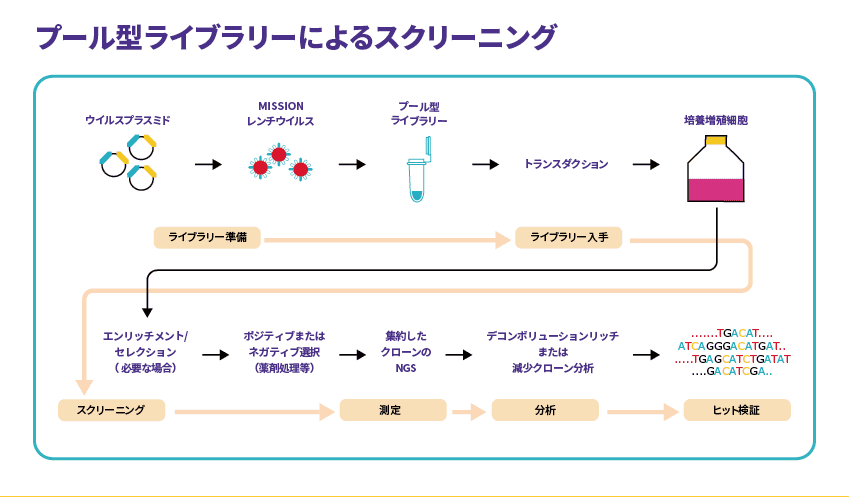
プール型のメリットは、セットアップや実験操作が比較的シンプルで、コストが抑えられる点です。そのため、遺伝子ターゲット毎のgRNA数を最大化することができます。in vivoスクリーニングも可能で、たとえばがん研究では細胞を個体に移植して、腫瘍化の有無でスクリーニングすることもできます*4。
しかし、データ解析にはNGSが必要で、解析に関する知識も要求されます。細胞導入にはレンチウイルス粒子のハンドリングが必須です。また、プロセス全体において十分な細胞数を確保するため、増殖速度の高い細胞を使う必要があります。
そのため、導入できる細胞は一般的に不死化細胞で、初代細胞は適していません。エンドポイントとするリードアウトは、細胞死や増殖能などに限られます。近年、シングルセル解析と組み合わせたスクリーニングも実施されています。詳細は10xGenomicsに適合するプール型CRISPRスクリーニングをご参照ください。
アレイ型
マルチウェルのマイクロプレートの各ウェルに、ターゲット遺伝子に対する1種類のgRNAが入ったタイプのライブラリーです(図2)。細胞にgRNAを導入してスクリーニング後、ウェルに含まれていたgRNAの情報と照らし合わせて対象遺伝子を決定します。
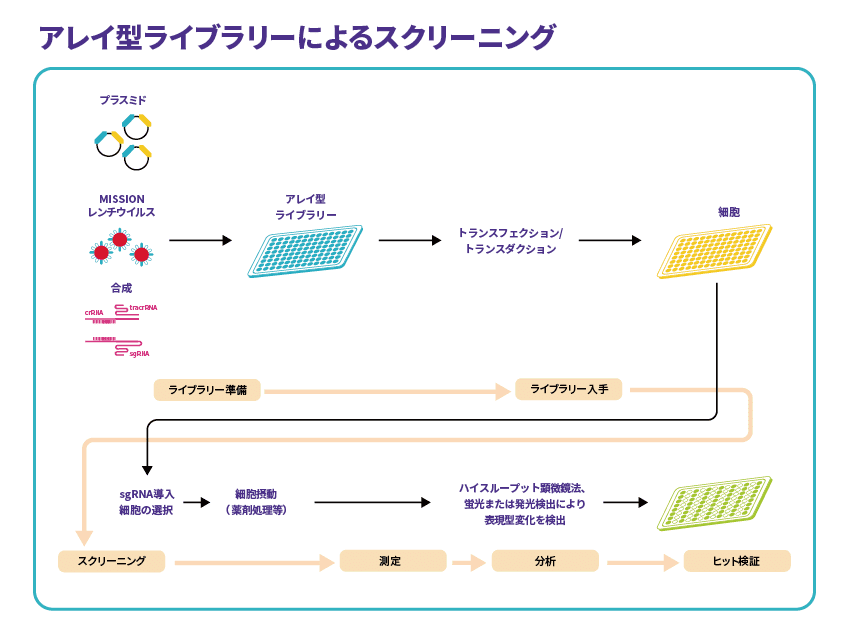
図2 アレイ型の原理
アレイ型のメリットは、NGSを必要とせず、遺伝子型と表現型の相関を直接解析できることです。ライブラリー導入において、増殖力の少ない初代細胞にも適応できます。また、レンチウイルスで感染させる以外にも、プラスミドDNAや合成RNAの状態で導入することもできます。リードアウトでは、蛍光や発光、形態学的変化など多様なオプションが利用可能です。
一方、セットアップは複雑で、ラボオートメーションのような特別な設備が求められます。クローン数が膨大なため、in vivoスクリーニングも難しくなります。クローン数に応じて作業時間が増え、プール型と比較してコストが割高になることにも注意が必要です。
表1は、プール型とアレイ型を比較したものです。利用できるリソースや目的に応じて最適なライブラリーのタイプを判断してください。
| プール型 | アレイ型 | |
|---|---|---|
| ライブラリー準備 | 1本のチューブに多数のgRNA | 各ウェルに1種類のgRNA |
| ライブラリー導入方法 | レンチウイルスのみ | レンチウイルス、プラスミド、RNA |
| スクリーニング所要時間 | 効率的な全ゲノムスクリーニング | クローン数に応じて作業時間増加 |
| スクリーニング方法 | in vivoスクリーニング可 | in vivoスクリーニング不可 |
| 分析 | NGSが必要 | NGS不要 |
| リードアウト | 細胞死や増殖などに限定(シングルセル解析と組み合わせることが可能) | 蛍光、ハイコンテント、生細胞イメージングなど多様 |
| 価格 | 低 | 高 |
プール型とアレイ型の特長は理解できたけれど…
「自分の研究テーマにはどちらが向いているのか」
「予算や導入方法の条件に合った提案がほしい」
そんな方は、ぜひこちらからお気軽にご相談ください。
CRISPR screeningのワークフロー
CRISPRスクリーニングのワークフローは、「事前の条件検討」「ライブラリー準備」「ライブラリー導入」「スクリーニング」「測定・分析」からなります。
1. 事前の条件検討
薬剤選択において導入されていない細胞を殺すための抗生物質濃度の決定、コントロールベクターを用いた感染多重度(MOI)の最適化などを行います。ベクターにCas9が含まれるオールインワンタイプを使わない場合には、Cas9発現細胞を作製する必要もあります。
2. ライブラリー準備
プール型ではレンチウイルスを作製するか、または市販されているレンチウイルスのプール型ライブラリーを用意します。アレイ型では、各ウェルに1種類のターゲット遺伝子に対するgRNAが既に振り分けられています。
3. ライブラリー導入
プール型ではレンチウイルスを感染させます。アレイ型ではレンチウイルスの感染、またはプラスミドDNAもしくは合成RNAの導入を行います。
4. 薬剤による導入細胞のセレクション・エンリッチメント
必要に応じて、抗生物質、もしくはGFPなどの蛍光マーカーを用い、導入細胞をセレクションします。
5. 目的のスクリーニング
薬物処理などの目的のスクリーニングを行います。プール型では個体に細胞を移植するin vivoスクリーニングも可能です*4。
6. 測定・分析
プール型の場合、スクリーニングした細胞を集約し、NGSによって測定・分析をします。アレイ型では生化学的、形態学的な変化を検出します。
ライブラリー選定ガイド
CRISPRライブラリーについて、弊社ではプール型とアレイ型の両方を取り揃えています。
プール型として、Feng Zhang博士およびMIT / Broad Instituteと提携したGeCKO全ゲノムライブラリーをはじめ、CRISPRaやCRISPRiを利用した全ゲノムライブラリーなどがあります。ベクターには、gRNAのみのものと、gRNAとCas9の両方が含まれるオールインワンタイプの2種類があります。
アレイ型は、Wellcome Sanger Instituteと共同で作成したSanger全ゲノムライブラリーがあります。フォーマットとしてレンチウイルスとグリセロールストックの2種類があります。Robotic handlingが可能な、1D, 2Dバーコード付きのプレートで提供されます(詳細はこちら)。ベクターにはBFPも含まれるため、蛍光によるセレクションも可能です。
※表のヘッダーをクリックしてソート、下のチェックボックスで列の表示/非表示を切り替えられます。
※表下部のスクロールバーにより横にスクロールできます。
| 種類 | ライブラリー | プール | クローン数 | 1遺伝子当たりの平均クローン数 | フォーマット | 仕様 | ベクター構成 |
|---|---|---|---|---|---|---|---|
| ノックアウト | Sanger ライブラリー |
アレイ型 | 〜40,000 | 2 | レンチウイルス | 10μL、> 1x106VP/mL in 約100枚 x 384ウェルプレート |
gRNAのみ、Puro、BFP |
| ノックアウト | Sanger ライブラリー |
アレイ型 | 〜40,000 | 2 | 大腸菌グリセロールストック | 50µL in 約400枚 x 96ウェルプレート |
gRNAのみ、Puro、BFP |
| ノックアウト | GeCKO2 ライブラリー |
プール型 | 〜124,000 | ~6 | レンチウイルス | 200μL(8x25 μL)、> 5x108VP/mL |
gRNAのみ または gRNA+Cas9、Puro |
| ノックアウト | Brunello ライブラリー |
プール型 | 〜76,441 | ~4 | レンチウイルス | 1mL (5 x 200μL)、> 5 x 108VP/mL |
gRNAのみ、Puro、BFP |
| 活性化 | SAM ライブラリー |
プール型 | 〜70,000 | ~3 | レンチウイルス | 200μL (8 x 25μL)、> 5 x 108VP/mL |
gRNAのみ、Puro または Zeo |
| 活性化 | Calabrese ライブラリー |
プール型 | 〜113,200 | ~6 | レンチウイルス | 200mL (5 x 200μL)、> 5 x 108VP/mL |
gRNAのみ、Puro |
| 阻害 | Dolcetto ライブラリー |
プール型 | 〜115,000 | ~6 | レンチウイルス | 1mL (5 x 200μL)、> 5 x 108VP/mL |
gRNAのみ、Puro、BFP |
| 阻害 | Whole Genome & Long Non Coding ライブラリー | プール型 | 〜258,000 | ~10 | レンチウイルス | 200μL (4 x 50μL)、> 1 x 108TU/mL |
gRNAのみ、Puro、BFP |
表2 弊社のヒトに対するCRISPR全ゲノムライブラリーの一例
その他製品ラインナップやプール型とアレイ型のワークフローの比較は、遺伝子スクリーニングページやカタログをご参照ください。カタログはカタログファインダーからダウンロードしてください。
\こんなことはありませんか?/
- どのライブラリーを選べばいいかわからない
- 実験の進め方が不安
もし一つでも当てはまったら、テクニカルスタッフのサポートが役に立つかもしれません。
オンライン技術サポートについて

専門スタッフが、お客様の研究目的や条件に最適なCRISPRライブラリーの選定をサポートいたします。
スクリーニングの進め方など技術的なご相談も承っておりますので、ぜひお気軽にお問い合わせください。
<References>
*1 Sredni ST, et al. Pediatr Blood Cancer. 2017;64(11). doi: 10.1002/pbc.26551.
*2 Pluvinage JV, et al. Nature. 2019 Apr;568(7751):187-192.
*3 Costa JR et al. Assay Guidance Manual [Internet]. Bethesda (MD): Eli Lilly & Company and the National Center for Advancing Translational Sciences; 2017
*4 Chow RD, et al. Trends Cancer. 2018 May;4(5):349-358.

下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須