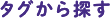CRISPR library —知っておきたい製品選定のコツ—

CRISPR-Cas9(クリスパー・キャスナイン)は代表的なゲノム編集ツールであり、全ゲノム遺伝子ライブラリーによるCRISPR screening(クリスパー・スクリーニング)にも利用されています。
CRISPR screeningに使用されるライブラリーをCRISPR library(クリスパー・ライブラリー)といいます。CRISPR libraryには多くの種類があり、どのライブラリーが自分の研究に適しているのか、悩むことも少なくありません。
当記事では、CRISPR libraryの特徴や選定基準などについて紹介します。
CRISPR library製品概要
CRISPR libraryと一口に言っても、ターゲット遺伝子やCRISPRの種類、製品フォーマットにより、数多くの製品が存在します。
図1は、Sigma-AldrichのCRISPR libraryをカテゴリ別に分けたものです。ご覧の通り、多くの製品を取りそろえています。
図1 Sigma-AldrichのCRISPR library製品。KO: ノックアウト、i: 干渉(ノックダウン、発現抑制)、a: 活性化(発現亢進)
全ゲノムを対象にしたライブラリーだけでなく、がん・アポトーシス関連遺伝子やキナーゼファミリー関連遺伝子に特化したライブラリーなどもあります。
まずは、全ゲノムを対象にしたいのか、特定の遺伝子群を調べたいのか、ターゲットの範囲を明確にしましょう。
テクニカルスタッフに相談する
※オンライン技術サポートも実施中
CRISPR libraryを用いたScreening原理
CRISPR-Cas9は、標的とするDNA配列を特異的に認識してDNAに結合するガイドRNA(gRNA)と、ヌクレアーゼであるCas9から構成されます。CRISPR-Cas9に関連する製品を以下のカタログで紹介しております。
多数の遺伝子をターゲットにしたgRNAを1つにまとめたものがCRISPR libraryであり、目的の表現型に関連する遺伝子を包括的に調べる事ができます。
CRISPR libraryには、大きく分けてプール型とアレイ型の2種類があります。
プール型は基本的にレンチウイルス粒子フォーマットになります。1本または複数のチューブに多数のレンチウイルス型gRNAが含まれており、レンチウイルス粒子を細胞に感染させgRNAを導入します。セットアップや実験操作が比較的シンプルでコストを抑えることができますが、次世代シーケンサー(NGS)によるデータ解析が必要です。
アレイ型は、マルチウェルプレートのウェルごとに1種類のgRNAが含まれており、標的遺伝子と表現型の相関を解析します。ウイルス感染だけでなく、プラスミドDNAや合成RNAの形態でも導入でき、NGS解析も不要です。その一方で、ロボットや特殊な設備が要求されます。
プール型とアレイ型の詳細な比較やCRISPR screeningの概要については、以下の記事をご覧ください。
CRISPR screeningとは?基礎やワークフロー、ライブラリー選択方法を解説
なお、ライブラリー製品によって、gRNAのみ用意されたタイプと、gRNAとCas9が1つのベクターに含まれるAll-in-oneのタイプがあります。
gRNAのみの製品を使う場合には、Cas9を同時に細胞導入またはウイルス感染させるか、事前にCas9を安定的に発現する細胞を作製する必要があります。
全ゲノムノックアウトライブラリー選択ガイド
ヒトを対象にした全ゲノムノックアウトライブラリーの製品ラインナップを表1に示します。
| ライブラリー | プール | フォーマット | gRNA数 | 1遺伝子あたりのgRNA数 | レポーター 遺伝子 |
コスト (製品番号) |
|---|---|---|---|---|---|---|
| Sigma | プール型 | レンチウイルス粒子 | 184,000 | 約10 | - | ++ (HWGCRISPR) |
| GeCKO2 | プール型 | レンチウイルス粒子 | 124,000 | 約6 | - | ++ (HGECKO2A) |
| Brunello | プール型 | レンチウイルス粒子 | 76,441 | 約4 | BFP | + (PCRISPR007) |
| Toronto | プール型 | レンチウイルス粒子 | 71,090 | 約4 | BFP | + (PCRISPR008) |
| Sanger | アレイ型 | レンチウイルス粒子 グリセロールストック |
34,332 | 約2 | BFP | ++++ HSANGERV(レンチウイルス粒子)、HSANGERG(グリセロールストック)) |
表1 プール型全ゲノムノックアウトライブラリー
Sigma全ゲノムライブラリーは1遺伝子あたりのgRNA数が多いのが特徴です。
Feng Zhang博士およびMIT / Broad Instituteと提携したGeCKO全ゲノムライブラリーはCas9がセットになったAll-in-oneタイプも用意しており、miRNAをターゲットとしたgRNAも含まれています。
これらよりもgRNAの数を抑えつつ、優れた特異性と正確性をもたせたのがBrunelloノックアウトライブラリーとTorontoノックアウトライブラリーV3です。
Brunelloノックアウトライブラリーは、MIT / Broad InstituteのJohn G Doenchらの報告をもとにライブラリーを最適化したもので、オンターゲット活性を最大化してオフターゲット活性を最小化していることが特徴です。
また、TorontoノックアウトライブラリーV3は、Jason MoffatらがCRISPR screeningの経験的データを用いてライブラリーを最適化したもので、スクリーニングの精度・効率の向上を目指したものです。
BrunelloとToronto ノックアウトライブラリーは、Sigma全ゲノムライブラリーやGeCKO全ゲノムライブラリーよりもさらにコストを抑えることができ、BFPによる蛍光セレクションが可能という特長もあります。
なお、プール型のGeCKO全ゲノムライブラリーについては、ヒトだけでなくマウスを対象にしたライブラリーもあります。All-in-oneタイプとgRNAのみの2種類があるので、Cas9発現細胞をご自身で用意できるかどうかによって製品を選択してください。
アレイ型を求める場合には、Sanger全ゲノムライブラリーがあります。ヒトおよびマウスの遺伝子に対して、それぞれ2種類のgRNAがあります。
レンチウイルス粒子フォーマットは約100枚の384ウェルプレートからなり、1gRNAあたり10 µL 、ウイルス力価は1x106 /mLです。また、グリセロールストックフォーマットも用意されており、約400枚の96ウェルプレートから構成されます。BFPによる蛍光セレクションや、グリセロールストックからPlasmidを精製し、TransposaseによるgRNA導入も可能です。
テクニカルスタッフに相談する
※オンライン技術サポートも実施中
CRISPRiとCRISPRaライブラリー
CRISPR-Cas9はDNA切断によるノックアウトが主な特徴ですが、Cas9を改変して発現抑制や発現亢進も可能になっています。
CRISPR-Cas9を活用した発現抑制はCRISPRi(CRISPR interference、CRISPR干渉)、発現亢進はCRISPRa(CRISPR activation、CRISPR活性化)とよばれます。
CRISPRiライブラリー
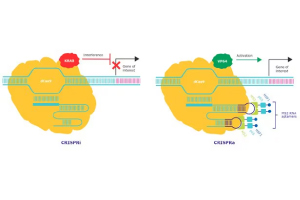
CRISPRiでは、Cas9に点変異を導入しヌクレアーゼ活性を欠失させたdCas9と、転写抑制因子であるKRABドメインを融合させたKRAB-dCas9を使用します(図2)。
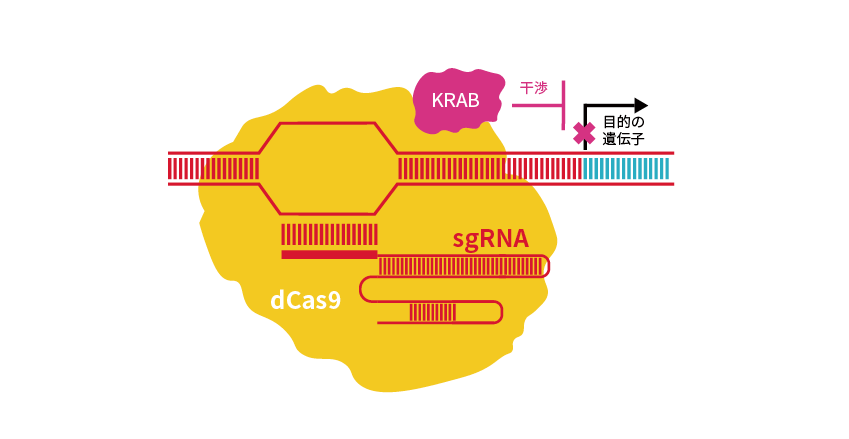
図2 CRISPRiの原理
表2は、Sigma-AldrichのCRISPRiライブラリーです。Dolcettoには遺伝子あたり約6個のgRNAが含まれています。Human Whole Genome & Non-Coding CRISPRi Library並びに各Pool製品には遺伝子あたり約10個のgRNAが含まれています(アルゴリズム上トップ5のgRNAのサブプールと、感度増加のための補足的な5個のgRNAのサブプール、および、ネガティブコントロールとしてのノンターゲットgRNAが含まれています)。
| ライブラリー | 遺伝子数 | コスト (製品番号) |
|---|---|---|
| Dolcetto (Whole) | 約18,000 | ++ (PCRISPR003) |
| Human Whole Genome & Non-Coding CRISPRi Library | 約18,000 | +++ (CRISPRILIB) |
| Druggable Genome Pool | 約2,500 | +(CRISPRIS01) |
| Cancer & Apoptosis Pool | 約3,000 | + (CRISPRIS02) |
| Stress Proteostasis Pool | 約3,000 | + (CRISPRIS03) |
| Mitochondria/Trafficking/Motility Pool | 約2,000 | + (CRISPRIS04) |
| Gene Expression Pool (Transcription Factors) | 約2,000 | + (CRISPRIS05) |
| Membrane Proteins Pool | 約2,500 | + (CRISPRIS06) |
| Unassigned Pool (Uncategorized Gene Function, Novel Targets) | 約2,000 | + (CRISPRIS07) |
| Common Cancer Cell Lines Pool & iPSC Pool: Common to 7 cell lines (5 cancer, iPSC, HFF) | 約500 | + (CRISPRIS08) |
| iPSC Pool | 約3,000 | + (CRISPRIS09) |
| Common Cancer Cell Lines Pool (HeLa, HEK293T, K562, MCF7, U87 cell lines) | 約1,500 | + (CRISPRIS10) |
※Dolcetto(Whole)のみ別途KRAB-dCas9が必要
表2 CRISPRiライブラリー
全ゲノムをターゲットにするCRISPRiライブラリーとして、KRAB-dCas9が付属するヒト全ゲノム・ノンコーディングCRISPRiライブラリーと、ご自身でKRAB-dCas9を用意するCRISPRi Dolcettoライブラリーがあります。
また、がん・アポトーシスプール、遺伝子発現プール(転写因子)や膜タンパク質プールなど、特定のパスウェイや領域に特化したサブプールもあります。ターゲットを絞ることでコストを大きく抑えることができます。
CRISPRaライブラリー
Sigma-AldrichのCRISPRaシステムは、dCas9にウイルス由来の転写活性化因子VP64を融合させたdCas9-VP64と、MS2-p65-HSF1活性化ドメインの2つの活性エフェクターを利用します。
gRNAにMS2 RNAアプタマーが含まれており、この2つの活性エフェクターと組み合わせることで遺伝子発現を促進します(図3)。
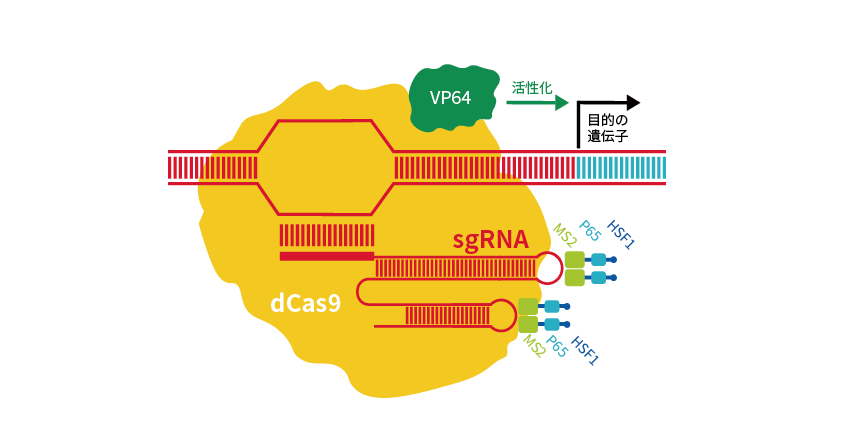
図3 CRISPRaの原理
表3は、Sigma-AldrichのCRISPRaライブラリーです。
| ライブラリー | フォーマット | gRNA数 | ターゲット 遺伝子数 |
1遺伝子あたりのgRNA数 | レポーター 遺伝子 |
コスト (製品番号) |
|---|---|---|---|---|---|---|
| SAM (Zeo) | レンチウイルス粒子 | 〜70,000 | 23,500 | 約3 | - | +++ (HSAMZEO) |
| SAM (Puro) | レンチウイルス粒子 | 〜70,000 | 23,500 | 約3 | - | ++ (HSAMPURO) |
| Calabrese | レンチウイルス粒子 | 〜113,200 | 〜18,885 | 約6 | - | ++ (PCRISPR001) |
表3 CRISPRiライブラリー
dCas9-VP64とMS2-P65-HSF1のレンチウイルス粒子が付属するSAMライブラリーと、これらの活性エフェクターが付属しないCalabreseライブラリーがあります。Calabreseライブラリーは、活性エフェクターをご自身で用意しなければなりませんが、その分コストが抑えられています。
まとめ
CRISPR libraryを使ったScreeningは規模の大きい実験になりますので、最適なライブラリー製品を選ぶことが重要です。
Sigma-Aldrichでは様々なライブラリー製品をご用意に加え、技術担当者による実験目的に合わせた製品提案が可能です。オンライン面談を含めた製品のご案内やご相談を承りますので、是非以下のフォームからお問い合わせください。
オンライン技術サポートのご紹介

全ゲノム遺伝子ライブラリーによるCRISPR screeningを実行する際には、念入りな情報収集と準備が必要です。
Sigma-Aldrich®では、オンライン面談も含めた製品のご案内やご相談を承っています。CRISPR screeningの詳細やお見積りをご希望の場合、以下よりお問い合わせください。
<References>
Metzakopian E, Strong A, Iyer V, Hodgkins A, Tzelepis K, Antunes L, Friedrich MJ, Kang Q, Davidson T, Lamberth J, Hoffmann C, Davis GD, Vassiliou GS, Skarnes WC, Bradley A.
Enhancing the genome editing toolbox: genome wide CRISPR arrayed libraries.
Sci Rep. 2017 May 22;7(1):2244. doi: 10.1038/s41598-017-01766-5. PMID: 28533524; PMCID: PMC5440395.
https://pubmed.ncbi.nlm.nih.gov/28533524/
Turner RJ, Golz S, Wollnik C, Burkhardt N, Sternberger I, Andag U, Cornils H.
A Whole Genome-Wide Arrayed CRISPR Screen in Primary Organ Fibroblasts to Identify Regulators of Kidney Fibrosis.
SLAS Discov. 2020 Jul;25(6):591-604. doi: 10.1177/2472555220915851. Epub 2020 May 19. PMID: 32425084; PMCID: PMC7309357.
https://pubmed.ncbi.nlm.nih.gov/32425084/

下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須