大腸菌におけるタンパク質発現とホスト選択 その種類と特徴

目的・課題に合わせたホスト選択
大腸菌の菌体内は動物細胞などと大きく環境が異なります。したがって、大腸菌でタンパク質を発現させる際には、正しくフォールディングが起こらず、タンパク質が不溶化してしまったり、正しく発現しなかったりといった問題が生じることがあります。これらの問題を解決するには、ベクター選択によるアプローチと、ホスト選択によるアプローチが考えられます。
この記事では、大腸菌におけるタンパク質発現を改善・向上させるホスト選択について、目的・課題別に紹介し、Origami™、Tuner™などの発現用ホスト株の特徴について解説します。
一般的なタンパク質発現にはBL21(DE3)やBL21が使用されます。また、動物由来原料不使用のVeggie™BL21(DE3)という選択肢もあります。目的タンパク質のラベリングを行う場合には、B834やB834(DE3)といったメチオニン栄養要求変異株ホストを使用します。
タンパク質の不溶化/不活性を改善したい場合、原因によって2つのアプローチが考えられます。まず、ジスルフィド結合の効率が悪く、正常にフォールドされたタンパク質が得られないときには、以下のtrxB/gorホストを用いて細胞質内でのタンパク質発現効率の改善を試みると良いでしょう。
- Origami 2
- Origami 2(DE3)
- Rosetta-gami™ 2
- Rosetta-gami 2(DE3)
- Rosetta-gami B
- Rosetta-gami B(DE3)
また、高レベルの発現誘導により正常にフォールドされたタンパク質が得られないときには、以下のlacY欠失ホストを用いてIPTG濃度依存型の誘導制御を試みましょう。
- Tuner
- Tuner(DE3)
- Rosetta-gami B
- Rosetta-gami B(DE3)
毒性タンパク質の発現によりタンパク質が全く発現しない、または大腸菌が増殖しない場合、以下のホストにより基底発現を抑制することで発現効率が改善することがあります。
- Tuner
- Tuner(DE3)
- NovaBlue
- NovaBlue(DE3)
- Any pLysS host
完全長のタンパク質が得られない場合は、大腸菌固有のコドン使用頻度の問題が考えられるため、レアコドン補充株の使用を検討してみましょう。
- Rosetta™
- Rosetta(DE3)
- Rosetta 2
- Rosetta 2(DE3)
- Rosetta-gami 2
- Rosetta-gami 2(DE3)
- Rosetta-gami B
- Rosetta-gami B(DE3)
- RosettaBlue™
- RosettaBlue(DE3)
インサート内の配列(繰り返し)の影響によりプラスミドが不安定になる場合、recA欠失ホストの使用によりプラスミドを安定化することが可能です。
- BLR(DE3)
- HMS174
- HMS174(DE3)
- NovaBlue
- NovaBlue(DE3)
ここからは、上記で紹介したホスト株のうち、Origami 2、Origami B、Rosetta-gami 2、Rosetta-gami B、Tuner (DE3) について、その特徴を解説します。
Origami 2 Host Strainsの特徴
K-12由来でチオレドキシンリダクターゼ(trxB)とグルタチオンリダクターゼ(gor)に変異をもつホストです。2種の還元酵素変異によって、細胞質をより酸化的な状態とし、ジスルフィド結合の形成を大幅に強化できます。その結果、必要なタンパク質が正しくおりたたまれた活性型で発現されやすくなります。カナマイシンもしくはアンピシリン耐性ベクターと併用できます。
遺伝子型は以下の通りです。
-
Origami 2(DE3) Genotype : Δ(ara-leu)7697 ΔlacX74 ΔphoA PvuII phoR araD139 ahpC galE galK rpsL F’[lac+ lacIq pro] (DE3) gor522::Tn10 trxB (StrR, TetR)
-
Origami 2(DE3)pLysS Genotype : Δ(ara-leu)7697 ΔlacX74 ΔphoA PvuII phoR araD139 ahpC galE galK rpsL F’[lac+ lacIq pro] (DE3) gor522::Tn10 trxB pLysS (CamR, StrR, TetR)
下の図は異なる条件下におけるpET-22bコンストラクトからのrRSC-cの発現を分析したものです。pET-22b/RSC-c組換えプラスミドを保有するBL21(DE3)およびOrigami(DE3)ホストは、OD600 0.6-1.0となるまで培養し、1 mM IPTGで発現誘導しました。培養液にIPTGを添加した後、37℃で4時間または20℃で24時間増殖を続けました。総タンパク質(T)、可溶性タンパク質(S)、および不溶性タンパク質(I)それぞれの画分のサンプルを調製し、15%SDS-ポリアクリルアミドゲルで分析した後、クマシーブルーで染色しました。
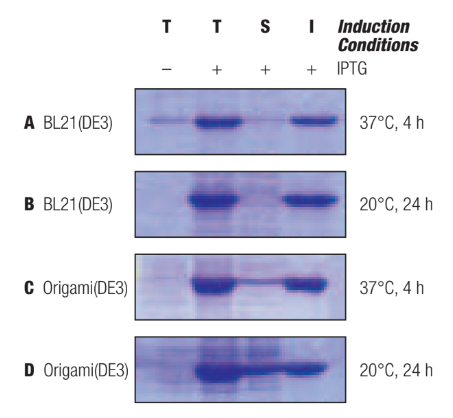
異なる条件下におけるpET-22bコンストラクトからのrRSC-cの発現
Origami B Host Strainsの特徴
IPTG濃度を調節することでタンパク質の発現レベルをコントロールできるBL21のlacY変異体(Tuner)に、ジスルフィド結合の形成を助けるチオレドキシンリダクターゼとグルタチオンリダクターゼの変異を加えたホストです。BL21のlonとompT欠損も受け継いでいるので、発現したタンパク質の安定性が上昇します。カナマイシン耐性株なのでカナマイシン耐性ベクターは使用できません。アンピシリン耐性ベクターと併用できます。
遺伝子型は以下の通りです。
-
Origami B(DE3) Genotype:F– ompT hsd SB(rB– mB–) gal dcm lacY1 ahpC (DE3) gor522::Tn10 trxB (KanR, TetR)
-
Origami B(DE3)pLysS Genotype:F– ompT hsd SB(rB– mB–) gal dcm lacY1 ahpC (DE3) gor522::Tn10 trxB pLysS (CamR, KanR, TetR)
Rosetta-gami 2 Host Strainsの特徴
発現タンパク質中に存在するジスルフィド結合の形成を助けるOrigami2(チオレドキシンリダクターゼ変異、グルタチオンリダクターゼ変異)に、大腸菌で存在量の少ないコドンに対応するtRNA(AUA、AGG、AGA、CUA、CCC、GGA、CGG)をコードするpRARE2を導入し、生物種差に関係ないタンパク質発現を可能にしたホストです。recA,endA,lacIq変異をもつK-12由来なので、細胞内でコンストラクトを安定に保持できます。カナマイシンもしくはアンピシリン耐性ベクターと併用できます。
遺伝子型は以下の通りです。
-
Rosetta-gami 2(DE3) Genotype:Δ(ara-leu)7697 ΔlacX74 ΔphoA PvuII phoR araD139 ahpC galE galK rpsL (DE3) F’[lac+ lacIq pro] gor522::Tn10 trxB pRARE2 (CamR, StrR, TetR)
-
Rosetta-gami 2(DE3)pLysS Genotype:Δ(ara-leu)7697 ΔlacX74 ΔphoA PvuII phoR araD139 ahpC galE galK rpsL (DE3) F’[lac+ lacIq pro] gor522::Tn10 trxB pLysSRARE2 (CamR, StrR, TetR)
Rosetta-gamiB Host Strainsの特徴
IPTG濃度を調節することでタンパク質の発現レベルをコントロールできるBL21のlacY変異体(Tuner)にジスルフィド結合の形成を助けるチオレドキシンリダクターゼ変異とグルタチオンリダクターゼ変異を加えた上、コドン補充プラスミドであるpRARE2を導入したホストです。Tuner、Origami、Rosettaの全ての特長を兼ね備えたドリームホストです。アンピシリン耐性ベクターと併用できます。
遺伝子型は以下の通りです。
-
Rosetta-gami B(DE3) Genotype:F– ompT hsdSB(rB– mB–) gal dcm lacY1 ahpC (DE3) gor522::Tn10 trxB pRARE (CamR, KanR, TetR)
-
Rosetta-gami B(DE3)pLysS Genotype:F– ompT hsdSB(rB– mB–) gal dcm lacY1 ahpC (DE3) gor522::Tn10 trxB pLysSRARE (CamR, KanR, TetR)
Tuner(DE3) Strain Competent Cellsの特徴
BL21のlacZY欠失変異体です。培養液中、全ての細胞のタンパク質発現レベルを調節できます。TunerのlacY変異が細胞内に取り込まれるIPTG量を均一にし、IPTG濃度依存的な誘導を行えます。IPTG濃度を調節することで、非常に低いレベルから充分に誘導したレベルまでタンパク質発現を制御できます。低いレベルでの発現は、発現が難しいタンパク質の可溶性や活性を促進することがあります。
遺伝子型は以下の通りです。
-
Tuner(DE3) Genotype:F– ompT hsdSB(rB– mB–) gal dcm lacY1 (DE3)
-
Tuner(DE3)pLysS Genotype:F– ompT hsdSB(rB– mB–) gal dcm lacY1 (DE3) pLysS (CamR)
| 製品名 | カタログ番号 | 包装単位 |
|---|---|---|
| Origami 2(DE3) Competent Cells | 71345-3 | 0.4 mL |
| 71345-4 | 1 mL | |
| Origami 2(DE3) Singles™ Competent Cells | 71408-3 | 11 rxn |
| 71408-4 | 22 rxn | |
| Origami 2(DE3)pLysS Competent Cells | 71346-3 | 0.4 mL |
| 71346-4 | 1 mL | |
| Origami B (DE3) Competent Cells | 70837-3 | 0.4 mL |
| 70837-4 | 1 mL | |
| Origami B (DE3)pLysS Competent Cells | 70839-3 | 0.4 mL |
| 70839-4 | 1 mL | |
| Rosetta-gami 2(DE3) Competent Cells | 71351-3 | 0.4 mL |
| 71351-4 | 1 mL | |
| Rosetta-gami 2(DE3)pLysS Competent Cells | 71352-3 | 0.4 mL |
| 71352-4 | 1 mL | |
| Rosetta-gami B (DE3) Competent Cells | 71136-3 | 0.4 mL |
| 71136-4 | 1 mL | |
| Rosetta-gami B (DE3)pLysS Competent Cells | 71137-3 | 0.4 mL |
| 71137-4 | 1 mL |
NovagenブランドのOrigami系ホスト
※ Origami 系のホストはpET-32、pET48b との併用で更なる効果が期待できます。これはチオレドキシン融合タグが細胞質中でのジスルフィド結合をさらに助けるためです。また,リダクターゼ変異の影響で培養時間が長くかかり18-24 時間程度必要です。
| 製品名 | カタログ番号 | 包装単位 |
|---|---|---|
| Tuner (DE3) Competent Cells | 70623-3 | 0.4 mL |
| 70623-4 | 1 mL | |
| Tuner (DE3) pLysS Competent Cells | 70624-3 | 0.4 mL |
| 70624-4 | 1 mL |
NovagenブランドのTuner系ホスト
以上、大腸菌におけるタンパク質発現を改善・向上させるホスト選択と、Origami系ホスト株やTunerなどの発現用ホスト株の特徴について解説しました。タンパク質発現に行き詰まったときには、現状の課題点を洗い出し、それを解決しうるホストを試してみましょう。また、タンパク質発現の改善にはベクター選択によるアプローチも可能です。両方の観点から検討すると良いでしょう。

下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須