2ステップRT-PCRのプロトコール
逆転写PCR(Reverse Transcription Polymerase Chain Reaction:以下RT-PCR)は、DNAを増幅するPCR技術をRNAにまで発展させたものです。2つのプロセスから成り、最初のプロセスではRNAの逆転写を行い、次のプロセスでDNAを増幅します。2つのプロセスをどのように行うかによって、RT-PCRには2つの方法があります。
逆転写に続いて、増幅をそのまま同じチューブで行うのが「1ステップRT-PCR」です。これに対して「2ステップRT-PCR」では、逆転写と増幅を別々のチューブで分けて行います。
この記事では、2ステップRT-PCRの詳細なプロトコールを2つご紹介し、実験を行う上での注意点、実際の実験例についても解説します。
無料PDF(PCR技術のすべて)はこちら
ニ次構造の多いものやGCリッチなRNAテンプレートを逆転写するためのプロトコール
RT-PCRの最初のステップである逆転写反応は、ターゲットとするRNAや配列の特性によって条件を調整する必要があります。ここでは、二次構造を多く含むものやGCリッチなRNAテンプレートに適した酵素の一例として「Transcriptor Reverse Transcriptase」(製品番号:3531317001/3531295001/3531287001)を用いたプロトコールをご紹介します。
この酵素は65℃以上の高温でも安定し、また反応が速いため、30分でcDNAの合成を行うことができます。14 kbまでの配列の転写が可能で、高温で反応させることができることから、二次構造やGCリッチな配列の逆転写にも最適な酵素です。
※すべての操作を始める前に、逆転写反応を行う温度になるようサーマルサイクラーを予熱しておきます。予熱温度は下記のステップ5をご覧ください。操作はすべてディスポーザブル手袋などを着用して行います。
-
試薬を準備します。凍結試薬を解凍し、各試薬はピペッティング操作により混ぜておきます。操作はコンタミネーションを避けて慎重に行ってください。試薬は氷上に置きます。
-
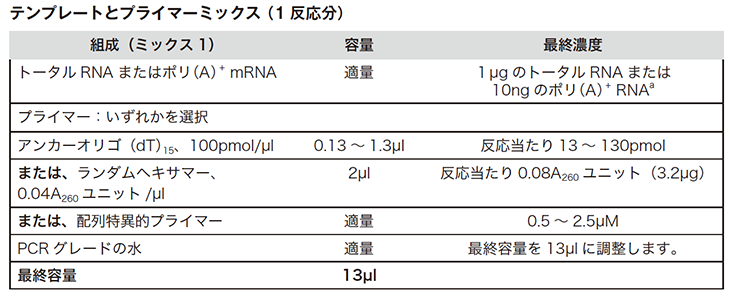
滅菌済みのヌクレアーゼフリー薄壁PCRチューブに、下記表に従ってテンプレートとプライマーの混合液を作ります。下記表を参照し、反応数に従って分量を調整してください。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p150)
-
(オプション)テンプレートが二次構造を多く含む場合などには、反応前に熱変性を行います。65℃で10分間反応させた後、すぐに氷上に戻します。
-
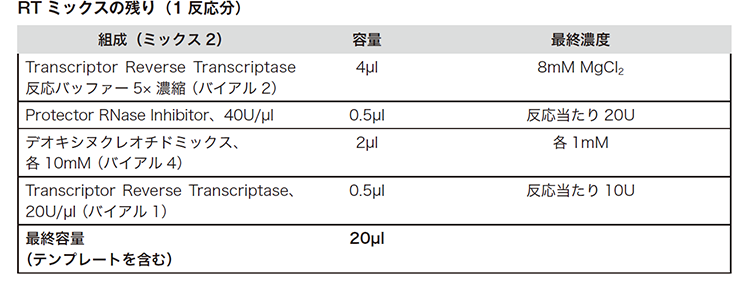
同じチューブに、下記表の試薬を上から順に加えます。下記表を参照し、反応数に従って分量を調整してください。泡立ちを避けるため、ボルテックスを使って攪拌しないでください。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p150)
-
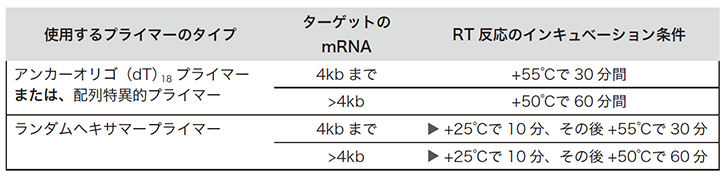
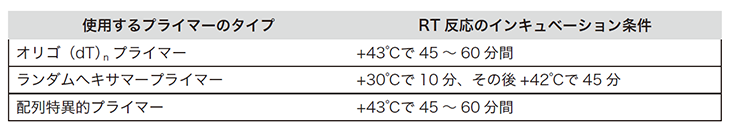
軽く遠心して混合液を底に集めた後、サーマルサイクラーに設置します。使用するプライマーやターゲットに応じて以下の条件で逆転写反応を行います。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p151)
-
既定のインキュベーション時間が終了した後、チューブを氷上に置き、反応を止めます。合成したcDNAをテンプレートとしてPCRを行い、cDNAを検出可能なレベルにまで増幅させます。
PCRのプロトコールについては、一般的なPCRとホットスタートPCRをご覧ください。
※PCRを行う前にcDNAを精製する必要はありません。1~5 μlの反応産物(cDNA)をPCRのテンプレートとして用います。初回実験では50 μlのPCR混合液に対して2 μlのcDNAテンプレート溶液を使うことをお勧めします。
完全長のcDNAを大量に合成するための逆転写反応プロトコール
RNase HはRNA-cDNA複合体のRNAを分解するため、RNase H活性を持つ酵素を使うと、完全長のcDNAが転写される前に、テンプレートRNAを失う可能性があります。
ここでは完全長cDNAや長い転写物を多く得たい場合に適した、RNase H活性を持たない酵素の一例として「ExpandTM Reverse Transcriptase」(製品番号:11785826001/11785834001)を使ったプロトコールをご紹介します。
※すべての操作を始める前に、逆転写反応に適した温度になるようサーマルサイクラーを予熱しておきます。予熱温度は下記のステップ5をご覧ください。操作はすべてディスポーザブル手袋などを着用して行います。
-
試薬を準備します。凍結試薬を解凍し、各試薬はピペッティング操作により混ぜておきます。操作はコンタミネーションを避けて慎重に行ってください。試薬は氷上に置きます。
-
滅菌済みのヌクレアーゼフリー薄壁PCRチューブで、下記表に従ってテンプレートとプライマーの混合液を作ります。下記表を参照し、反応数に従って分量を調整してください。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p152)
-
逆転写反応前に熱変性を行います。65℃で10分間反応させた後、すぐにチューブを氷上に戻します。
-
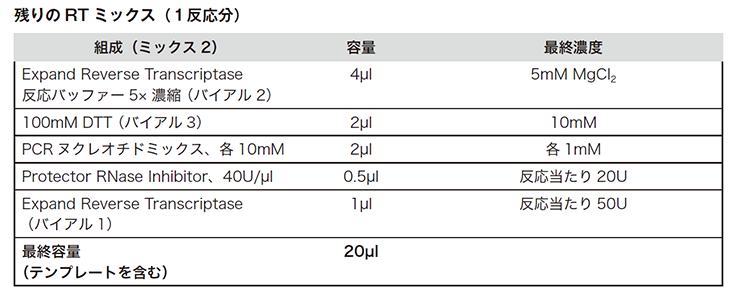
同じチューブに、下記表の試薬を上から順に加えます。下記表を参照し、反応数に従って分量を調整してください。泡立ちを避けるため、ボルテックスを使って攪拌しないでください。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p152)
-
軽く遠心して混合液を底に集めた後、サーマルサイクラーに設置します。使用するプライマーやターゲットに応じて以下の条件で逆転写反応を行います。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p153)
-
既定のインキュベーション時間が終了した後、チューブを氷上に置き、反応を止めます。合成したcDNAをテンプレートとしてPCRを行い、cDNAを検出可能なレベルにまで増幅させます。
PCRのプロトコールについては、一般的なPCRとホットスタートPCRをご覧ください。
※PCRを行う前にcDNAを精製する必要はありません。1~5 μlの反応産物(cDNA)をPCRのテンプレートとして用います。初回実験では50 μlのPCR混合液に対して2 μlのcDNAテンプレート溶液を使うことをお勧めします。
※合成したcDNAをすぐに使わない場合には、以下の表の方法に従って保存してください。

(出典元:Roche PCRアプリケーションマニュアル 第3版 p153)
逆転写反応のトラブルシューティング
RT-PCRが成功しない原因はいくつか考えられます。その1つとして、逆転写反応がうまく進まなかったためにcDNAが合成されていない可能性があります。
cDNAは、アガロース電気泳動により、クオリティおよびサイズを確認することができます。ターゲットとした配列のcDNAが得られていないことが確認できた場合には、以下の項目を検討してください。
-
トータルRNAを使っている場合には、反応液中のテンプレートRNAの量を全量1 µgまでの範囲で増やすか、ターゲットとなる配列の増幅効率を上げるため、mRNAを精製してテンプレートとします。
-
抽出過程で用いるエタノールやフェノール、SDSなどの反応を阻害する化学物質が逆転写反応を妨げている可能性があります。これらの化学物質がテンプレート溶液に残らないように、完全に除去することも大切です。
-
テンプレートとなるRNAの精製度をアガロースゲル電気泳動で確認します。mRNAであれば500 bp~8 kbの間でスメア状に見えます。大部分は1.5~2 kbに分離します。
真核生物のトータルRNAの場合には、18Sと28SのrRNAの明瞭なバンドが確認できます。このようなバンドが確認できない場合は、RNA分解が起こっている可能性がありますので、完全にRNaseフリーになるよう実験系を再構築してください。 -
逆転写用プライマーとしてオリゴdTやランダムヘキサマーを使っている場合には、配列に特異的なプライマーに変えることをお勧めします。また、配列特異的プライマーは、mRNAに(アンチセンスの方向に)結合する配列であることを確認してください。
-
逆転写反応のインキュベーション時間を長くしてください。インキュベーション時間を長くして、低い温度で反応させることで、完全長のcDNAの収量が高くなります。
逆転写反応後のPCRステップでの注意点
RT-PCRでの逆転写に続く増幅反応は、通常のPCR反応と同様ですが、いくつか注意点があります。
- cDNAテンプレートの容量がPCR反応液の全量の10%を超えないようにしてください。cDNAの量が多いと、PCR反応が阻害されることがあります。
- cDNAを含む溶液にはMg2+イオンがすでに含まれています。溶液にMg2+を加える場合には、すでに含まれている量も含めて、反応液全体の濃度が適当になるようにしてください。
RT-PCRの実験例
実験例として、ジストロフィン遺伝子からさまざまな長さの配列をターゲットとして増幅した結果をご紹介します。
最初の逆転写のステップでは、ヒト筋肉由来のトータルRNA(1 μg)をテンプレートとして、ヒトジストロフィン遺伝子配列に特異的な複数のプライマーを使って、「Expand Reverse Transcriptase」(製品番号:11785826001/11785834001)でcDNAを合成しました。
次に、未精製のcDNAを5 μlずつに分け、それぞれに異なる長さの配列を増幅するためにデザインしたプライマーセットで、「ExpandTM Long Template PCR System」(製品番号:11681834001/11681842001/11759060001)を用いてPCRを行いました。
各PCR産物の一部をアガロースゲル電気泳動で確認した結果が下図です。両端のレーン1と11はDNA分子量マーカーII、レーン2と10は分子量マーカーXです。ターゲットとした配列の長さは以下の通りです。
レーン3:1,857 bp、レーン4:4,041 bp、レーン5:5,893 bp、レーン6:7,678 bp、レーン7:9,556 bp、レーン8:11.9 kb、レーン9:13.5 kb
アガロースゲル電気泳動の写真から、各レーンに目的とした長さのバンドが確認できます。(図1)
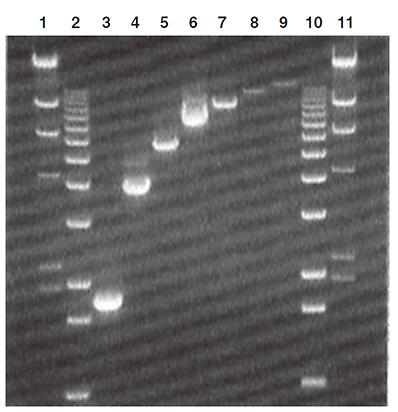
図1 ジストロフィン遺伝子から13.5 kbまでの長さの配列を2ステップRT-PCRで増幅した結果(出典元:Roche PCRアプリケーションマニュアル 第3版 p155)
以上、RT-PCRの詳細なプロトコールと注意点などをご紹介しました。PCR実験は、テンプレートの特性や増幅したい配列の長さ、実験の特性により大きく条件が変わります。RT-PCRを行う際の条件検討の参考にしていただければ幸いです。
<参考文献>
Degen, Hans-Joachim, Ph.D. 他. PCRアプリケーションマニュアル. 第3版, Roche Diagnostics GmbH, Mannheim, 2006
<無料PDFダウンロード> PCR技術のすべて
このカタログでは、PCRの歴史やさまざまなPCRの原理、さらにはトラブルシューティングなど、PCRに関連する便利な情報を掲載しております。
▼こんな方にオススメ
・PCRを基礎から学びたい方
・PCR実験に悩んでいる方
・qPCR条件の最適化について知りたい方

下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須