DIGシステムを用いたDNAの標識手法(ラベリング)

DIGシステムを用いた核酸の標識
DIGはステロイドハプテンであるDigoxigenin(ジゴキシゲニン)を用いることでRI(放射性同位元素)を使用せずに、核酸を効率的に標識・検出することができるシステムです。Non-RI DIGシステムはRIに比べて安全性が高いこと、高感度、短い露光時間で検出が可能であるなど、多くの利点を持っており、サザンブロットなどのフィルターハイブリダイゼーションやin situハイブリダイゼーション (ISH法) に活用されています。
このシステムはDNA、RNA、オリゴヌクレオチド、いずれの核酸の標識にも使用することができますが、この記事では特にDIGを用いてDNAを標識する方法や注意点について紹介します。
DIGを用いたDNAの標識手法は次の3種類があります。
- PCRによる標識 (PCRラベリング)
- ランダムプライミングによる標識
- ニックトランスレーションによる標識
以下で、具体的に見ていきましょう。
PCRによるDNAの標識とメリット
PCRラべリングは、テンプレートが極めて少量あるいは短いといった制限がある場合に有効な手法です。この手法は他のラべリング方法より至適化が容易なうえ、標識プローブを高い収量で産出できるというメリットがあり、もっとも簡単なDNAの標識方法です。
PCRラべリングでは、DIG-dUTPを組み込まれた熱安定性ポリメラーゼがテンプレートDNAの特定領域を増幅します。その結果、特異性と感度ともに優れたラべリング用ハイブリダイゼーションプローブが産生されます。
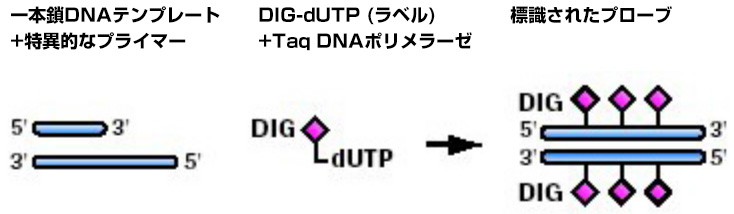
通常のPCR反応を通じてDigoxigenin-11-dUTPが、新しく合成されるDNAに取り込まれます。PCRによってごく微量のDNAもµgオーダーにまで増幅が可能です。PCRラべリングの注意点としては、あらかじめターゲットのシークエンス情報が必要となり、それに対する特異的なプライマーをカスタムで合成する必要があることです。
ジゴキシゲニンで標識されたdUTPは、DNAポリメラーゼを使用した酵素的な核酸合成により、 プローブに容易に取り込まれます。Non-RIラべリングとPCR反応は、PCR産物の分析および(限られた標的配列からの)標識プローブ調製のための強力なコンビネーションツールです。
PCRによるDNAの標識のヒント
PCR条件:PCR増幅のパラメータ(サイクル設定、テンプレート濃度、プライマー配列およびプライマー濃度)を至適化しましょう。至適化は、各テンプレートおよびプライマーごとに、DIG-dUTP不在下で行います。
テンプレート:クローンを挿入したプラスミドDNAから切り出したターゲットのDNA配列をテンプレートとして用いると良い結果が得やすいです。反面、ゲノムDNAは扱いが困難です。テンプレート濃度は、プローブの特異性に関して大きな影響を与えるファクターとなります。
ラベリング:ロシュのPCR DIG Probe Synthesis Kitを使用すると、他のラべリング手法に比べて至適化のプロセスが極めて容易となります。その理由としてExpand High Fidelity PCR Systemが採用されていることが挙げられます。このPCR酵素は以下の特長を持っています。
- GCリッチな領域もテンプレートとして使用できる。
- ほとんどのテンプレートに対して標準的な濃度のMgCl2(1.5mM)を適用できる。
したがって、至適化のプロセスが必要なのはキット中のdNTPストック溶液(10x)によるDIG-dUTP濃度の調整だけになります。この調整が必要な理由は、DNAテンプレートによっては(特にGC含有量が多い、またはテンプレートが長い場合)標準的なDIG-dUTP濃度においても、十分な増幅効率が得られない場合があるためです。
ランダムプライミングによるDNAの標識とメリット
ランダムプライミングでは、テンプレートの長さはほとんど問題になりません。ただし、ごく短い配列に対しては、PCRによるDNAの標識の方が適しています。
標識プローブはサザンブロットによるシングルコピーの遺伝子検出に最適ですが、もちろん通常のドットブロット、ノーザンブロットにも使用することができます。
プライマー自体が異なる6塩基の配列を有するため、プローブ産物もまちまちな長さをもつ断片となります。したがってゲル上でも固有のバンドではなく、スポット状のシミとなって検出されます。標識プローブ長の分散は、元となるテンプレート長に依存しています。
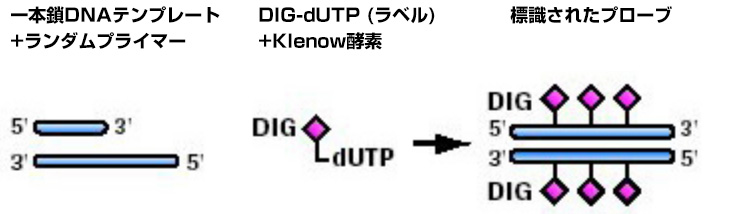
ランダムプライミングに用いられるKlenow酵素(5’→3’エキソヌクレアーゼ活性を失ったDNAポリメラーゼ I)は6量体プライマーとアルカリ不安定DIG-11-dUTP存在下で、DNAテンプレートを複製します。平均的に、Klenow酵素は20~25ヌクレオチド配列毎にDIG部位を挿入します。ラベル産物は0.10~0.03pgのターゲットDNAを検出できる、均一かつ高感度なハイブリダイゼーションプローブとなります。
ランダムプライミングではDIG分子の配列が重要となるので注意しましょう。これは、DIG分子同士が近すぎた場合、立体障害によってAnti-DIG抗体が標識プローブと結合できなくなる場合があるためです。
ニックトランスレーションによるDNAの標識とメリット
ニックトランスレーション法は、Rigby [1]によって提唱されたのち、Langer [2]によって核酸のアナログを取り込む用途へと進化を遂げました。
ここで述べる手法では、DNA産物のおおよそ20~25塩基配列毎に修飾されたヌクレオチド(DIG-、Biotin-、Fluorescein-、またはTetramethylrhodamine-dUTP)が挿入されることになります。このラベル密度は、修飾ヌクレオチドを酵素が取り込むうえで効果的なだけでなく、免疫染色による検出(間接法)において最も高感度なターゲットを産出することができます。in situハイブリダイゼーションに使用する場合、ニックトランスレーションで作成できる適正なラベル断片長はおよそ200~500ベースです。ニックトランスレーション法ではDNAの変性は必要ありません。
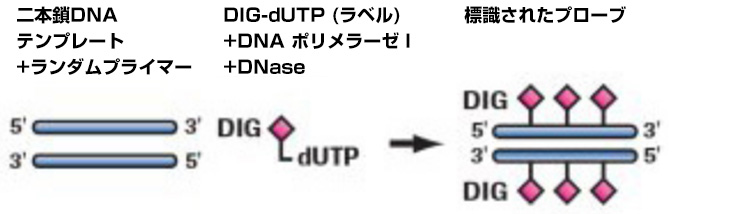
E.coliポリメラーゼ IとDNase IをdsDNAテンプレートに添加すると、ニックトランスレーション反応によって、合成されるDNAにはDigoxigenin-11-dUTPが取り込まれるようになります。ニックトランスレーションは核酸のin situラべリングに極めて適した手法です。
以上、DIGシステムによってDNAを標識する方法について紹介しました。DNAの検出感度や手法について悩んでいる方はぜひ一度、DIGシステムを検討してはいかがでしょうか。また、DIGシステムはRNAやオリゴヌクレオチドの標識にも使用できます。用途に合わせてこれらも検討してみてください。
References
[1] Rigby, P. W. J.; Dieckmann, M.; Rhodes, C.; Berg, P. (1977) Labeling deoxyribonucleic acid to high specific activity in vitro by nick translation with DNA polymerase I. J. Mol. Biol. 113, 237–241.
[2] Langer, P. R.; Waldrop, A. A.; Ward, D. C. (1981) Enzymatic synthesis of biotin-labeled polynucleotides: Novel nucleic acid affinity probes. Proc. Natl. Acad. Sci. USA 78, 6633–6637.
下記フォームでは、M-hub(エムハブ)に対してのご意見、今後読んでみたい記事等のご要望を受け付けています。
メルクの各種キャンペーン、製品サポート、ご注文等に関するお問い合わせは下記リンク先にてお願いします。
*入力必須